Gerade Biologen/Biotechnologen ohne Vorkenntnisse aus der Informatik, haben oft Probleme beim Verständnis grundlegender Verfahren der Bioinformatik. In einer kleinen Serie von Tutorials, werde ich versuchen, dieser Zielgruppe den Einstieg zu erleichtern. Die Codebeispiele folgen der Syntax der Programmiersprache C#, die abstrakten Verfahren können aber auf andere objektorientierte Programmiersprachen übertragen werden. (z.B. Java, C++, VisualBasic…)
Die Theorie zu lesen ist zwar schön und gut, doch am Besten verstehen, was vor sich geht kann man, indem man es selbst macht. Ich empfehle jedem, die Codebeispiele nachzubauen, bzw. in echt nachzuvollziehen! Keine Sorge, du musst nicht alles selbst machen. Im folgenden findest du zunächst eine Anleitung, wie du dir die notwendige Software installierst. Anschließend kannst du ein bereits vorbereitetes Projekt herunterladen.
- Einführung in die Bioinformatik
- Scoring von Alignments
- Der Needleman-Wunsch-Algorithmus
- Die F-Matrix (abstrakt)
- Auswählen des Alignments (abstrakt)
- F-Matrix (Code)
- Auswählen des Alignments (Code) — coming soon
Vorbereiten der Software
Um die Codebeispiele testen zu können, benötigst du eine sogenannte ‘Entwicklungsumgebung’. Bei “Microsoft Visual Studio Express 2012 for Windows Desktop” handelt es sich um eine kostenfreie Version von Visual Studio 2012. Als Student bekommt man über MSDNAA übrigens auch eine kostenfreie Lizenz für die Vollversion, welche sonst über 600€ kostet!
Herunterladen kannst du die Software hier: http://www.microsoft.com/visualstudio/eng/downloads#d-express-windows-desktop
Wer noch mit Windows XP/Vista ‘arbeitet’ sollte 1. Visual C# 2010 Express als Übergangslösung verwenden und 2. Upgraden!
Ich empfehle die englische Version, da die deutschen Übersetzungen der Begriffe ziemlich nichtssagend sind und bei der Suche nach Problemlösungen im Internet nur im Weg sind.
Mit einem Doppelklick öffnest du den Installer. Der macht den Rest dann von alleine.

Die Installation dauert eine Weile – Visual Studio ist schließlich eine ziemlich mächtige Software und mit der Version die du gerade installierst, kann man jede Art von Desktop-Anwendungen erstellen.
Nachdem die Installation fertig ist, kann du das bereits vorbereitete Beispielprojekt herunterladen: https://www.dropbox.com/s/x06vcugtg3yvjsj/Bioinfo01.zip?dl=0
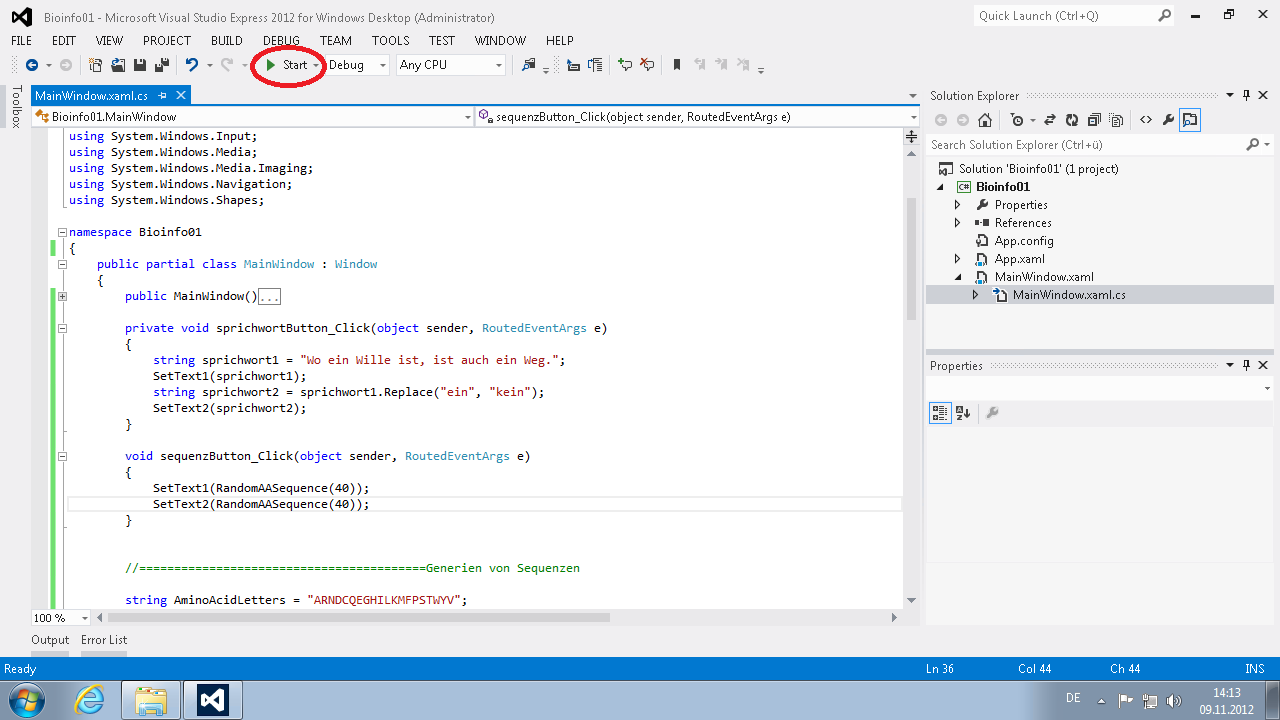
Entpacke das Archiv an einen Ort deiner Wahl. Im Ordner ‘Bioinfo01’ befindet sich die Datei ‘Bioinfo01.sln’. Öffne sie mit einem Doppelklick. (Die Aufforderung dich zu registrieren kannst du erstmal wegdrücken.)
Als nächstes sollte es etwa so aussehen:
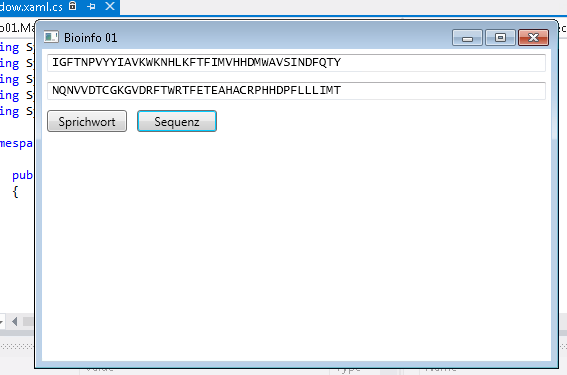
Mit einem Klick auf “Start” kannst du das Programm starten:
Wie das Programm funktioniert erkläre ich gleich, doch zunächst brauchen wir ein paar…
Grundlagen der objektorientierten Programmierung
Zum vereinfachten Verständnis unterscheiden wir zunächst drei Dinge:
- Klasse – definiert die ‘Art’ eines Objekts, zum Beispiel string, int, oder double
- Instanz – das Objekt selbst (repräsentiert eine Information)
- Funktion – führt eine Operation durch (zum Beispiel text.Replace(‘A’, ‘B’) macht aus jedem ‘A’ in der string-Instanz ‘text’ ein ‘B’)
Daneben gibt es noch die sogenannte ‘Syntax’, welche festlegt, wie Code zu interpretieren ist. (Vergleichbar mit Grammatik in der Sprache.)
Ein einfaches Codebeispiel – manipulieren eines strings

In Zeile 1 wird zunächst ein string mit der Bezeichnung ‘beispielText’ initialisiert. Ihm wird die Zeichenkette “Wo ein Wille ist, ist auch ein Weg.” zugewiesen.
Anschließend wird die Methode ‘Replace’ auf das Objekt ‘beispielText’ angewandt und der zurückgegebene Wert der dem Objekt ‘beispielText’ zugewiesen.
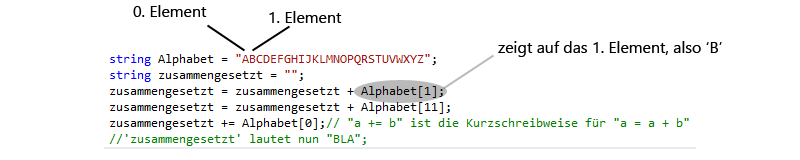
Referenzieren einzelner Zeichen in einem string
Um eine Zeichenkette (Aminosäuresequenz) zu analysieren, reicht es nicht, sie als Ganzes anzuschauen. Man muss auch auf einzelne Zeichen in der Zeichenkette zugreifen können.
Hierfür wird die Zeichenkette als sogenanntes Array interpretiert. Ein Array hat eine definierte Länge und mit einer Ganzzahl (int) kann auf seine Elemente zugegriffen werden.
Nachdem ‘Alphabet’ und ‘zusammengesetzt’ initialisiert wurden, wird in Zeile 3 zusammengesetzt’ ein neuer Wert zugewiesen. Der neue Wert setzt sich zusammen aus dem vorherigen Wert von ‘zusammengesetzt’ und dem 1. Element aus ‘Alphabet’.
In der nächsten Zeile wird ‘zusammengesetzt’, welches jetzt den Wert “B” hat, der Wert “B” + ‘L’ zugewiesen.
Die dritte Zeile macht genau das selbe, nur wird hier die Kurzschreibweise benutzt. Dies ist in vielen Fällen übersichtlicher!
Generieren einer zufälligen Aminosäuresequenz
Jede Aminosäure wird durch ihren Ein-Buchstaben-Code repräsentiert. Dadurch kann eine Aminosäuresequenz als string behandelt werden.
Zunächst definieren wir einen string, welcher alle Ein-Buchstaben-Codes enthält:
string AminoAcidLetters = “ARNDCQEGHILKMFPSTWYV”;
Die Bedeutung der Zeichen kann dieser Tabelle entnommen werden.
Damit wir uns auch an anderer Stelle einfach zufällige Sequenzen erzeugen können, schreiben wir den Code als neue Methode. Die Methode bekommt einen Parameter vom Typ ‘int‘ mit der Bezeichnung ‘length’. Dadurch kann beim Aufruf der Methode die Länge der zu generierenden Sequenz festgelegt werden. Die Methode heißt ‘RandomAASequence’ und gibt einen string zurück.

Um effizientes Arbeiten auch bei sehr langen Sequenzen sicherzustellen, verwenden wir die StringBuilder Klasse an Stelle der string Klasse. Mehr Informationen zum Unterschied zwischen string und StringBuilder findest du hier.
Besonders wichtig ist, dass du verstanden hast, was in Zeile 35 vor sich geht: Bei “r<lenght” handelt es sich um ein sogenanntes Statement. Es kann entweder wahr (true) oder falsch (false) sein. Alles was in den Klammern (Zeile 36-41) steht wird immer wieder ausgeführt, bis das statement ‘r<length’ nicht mehr wahr, also false ist. Dieser Fall tritt ein, wenn die Variable ‘r’ den Wert von ‘length’ oder größer annimmt. Damit wir uns nicht in einer endlosen Schleife verfangen, müssen wir mit jeder Iteration ‘r’ um 1 erhöhen!
Nachdem die for-Schleife ausgeführt wurde, enthält ‘sequence’ unsere zufallsgenerierte Aminosäurensequenz. In Zeile 43 wird diese an den Aufrufer (caller) der Methode zurückgegeben.
Der oben Dargestellte Code ist durch die vielen Kommentare leider etwas unübersichtlich geworden. Gekürzt und optimiert sieht er so aus:
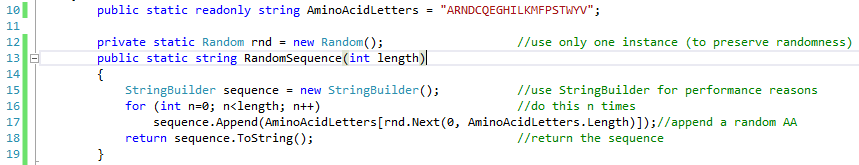
Das Aufrufen von RandomSequence(40) gibt nun eine zufällige Sequenz von 40 Aminosäuren zurück. Zum Beispiel: GHHFWSPMFGLKMNKMWHYNDGYFGHEHPAYHFPRRWCPF
Anders als das ausführliche Codebeispiel, ist dieses noch nicht im Bioinfo01-Beispielprojekt. Um ein Gefühl dafür zu bekommen, kannst du es ja mal nachprogrammieren 😉
Wie wichtig die Verwendung des StringBuilders anstatt eines normalen strings ist, zeigt der direkte Vergleich: die Generierung einer 100 000 Zeichen langen Sequenz dauert bei Verwendung des string Typs etwa 300 mal so lang!
Als Nächstes geht es weiter mit dem Scoring von Alignments.

okan duygun
Lieber Cake,
die Adresse : http://dl.dropbox.com/u/7813771/Blog/Bioinformatik/Bioinfo01.zip, ist aufgehoben oder versetzt, ich kann die Datei nicht öffnen.n ICh bitte um informastion , danke im Vorraus
theCake Post author
Hallo, danke für den Hinweis. Ich habe die links aktualisiert: https://www.dropbox.com/s/x06vcugtg3yvjsj/Bioinfo01.zip?dl=0
okan duygun
Sorry , daß ich dich nerve, aber der Pfad lässt sich bei mir im Studio 2012 nicht öffnen, es
folgt error : Die Projektdatei konnte nicht geladen werden. Ein Teil des Pfades “C:\Users\Win7 Pro x64\AppData\Local\Temp\Temp3_Bioinfo01.zip\Bioinfo01\Bioinfo01\Bioinfo01.csproj” konnte nicht gefunden werden. C:\Users\Win7 Pro x64\AppData\Local\Temp\Temp3_Bioinfo01.zip\Bioinfo01\Bioinfo01\Bioinfo01.csproj
Vielen Dank, für die Antwort
theCake Post author
Versuche mal die zip-Datei richtig zu entpacken (nicht nur im temporären Ordner)